

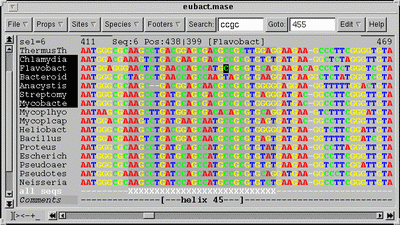
图形化多序列比对编辑器,能够读各种比对格式(MSF, CLUSTAL, FASTA, PHYLIP, MASE),可手动 列. 详细介绍

『RTF_New』(需等宽字体方可在Word显示正常),输入格式推荐用ALN格式,用记事本打开Clustalx比对文件,复制全部内容粘贴在相应选项框内. 相关教程详见本人的《序列着色软件Boxshade图解教程(By Raindy)》,百度文库可下载:


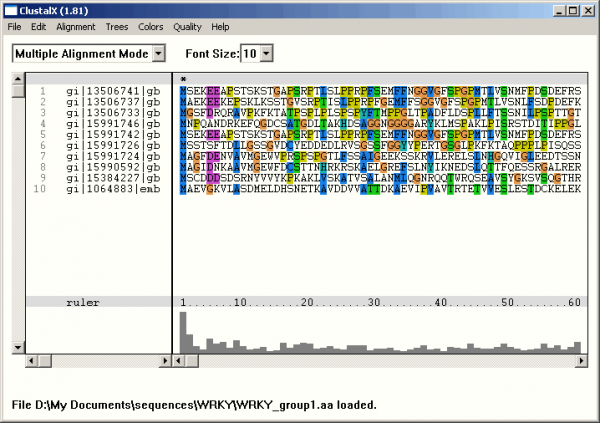
我有4个功能相似的蛋白质序列,都含有多铜离子结合区域,想通过软件比对后,显示出他们之间的保守区域,也就是铜离子结合区域.如下面给出的例子! 想请教各位大侠使用何软件才能做到,谢谢!

,默认打开SeqPage,此时点击菜单上的\"File\"-\"Open\"-选择上步保存的fasta文件,默认所有序列被选中.对于待分析的序列组,建议逐个选择进行分析,如下图的F组,可以先选定FJ|QK44. ,如下图,红色圈处为参考序列组,蓝色为待分析的P

3、点ALIGNMENT,在默认alignment parameters下,点击Do complete Alignment .在新出现的窗口中点击ALIGN进行比对,这时输出两个文件(默认输出文件格式为Clustal格式):比对文件test.aln和向导树

2.3 杏野黑樱苷水解酶预测蛋白及分析 使用 NCBI 中 ORF Finder 软件对序列分析(图 6,图 7) ,PaLTPH CDS 序列和 PaMJPH CDS 序列包含起始密码子和终止密码子,为全长的开放阅读框 ORF(open readin
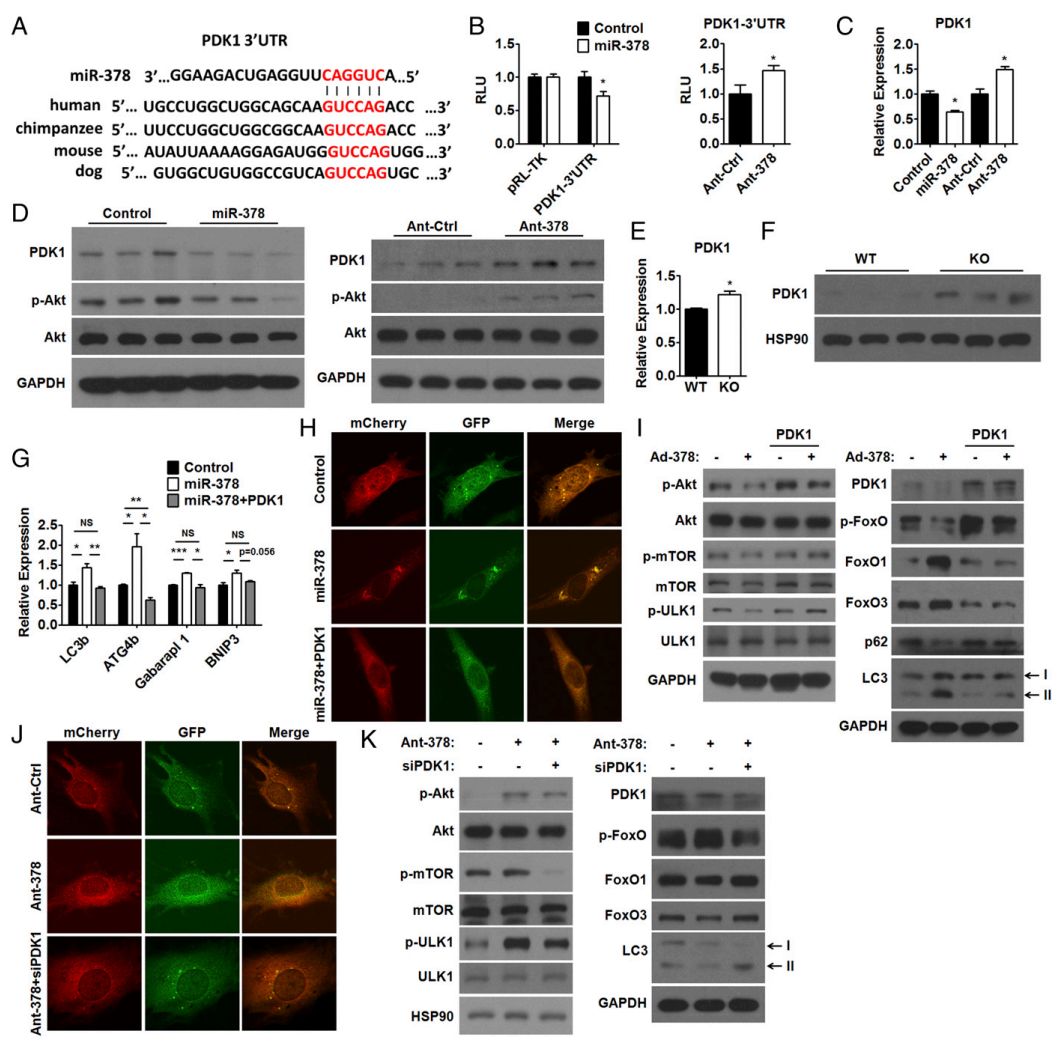
是miR- 378和PDK1-3′UTR从不同的物种进行序列比对; 通过荧光素酶分析显示,转染miR-378与转染抗miR-378者对荧光素酶的表达均有影响,即从机制的水平说明miR-378与PDK1可直接相互作用; 而 H+-ATP 酶是溶酶体

这里需要用到两个软件,一个是Clustal X,这个就不用说了.这时相信你已经有了clusalX 序列比对 后的结果,有两个文件,一个是.aln文件,存放多 序列比对 的结果.一个是.dnd文件,根据需要,可以转为系统发育树图,这个就不在今

该软件要求您的电脑要装有Java环境(进入下载jre.Java环境)才能正常使用 为用Java语言写的多序列比对编辑器,是一个专业的CAD程序.专门用于显示、分析和跟踪多个序列对齐.使用它来查看和编辑序列排列,用系统树和主成分分析(PCA)来分析它们,

Clustalw的使用(一) Clustalw还提供了命令调用形式的使用方式,方便于批处理过程,下面是一个典型的执行多序列比对的clustalw命令: 第三步:开始序列比对. 第四步:比对完成,选择保存结果文件的格式
Clustal的工作原理 Clustal输入多个序列>>>快速的序列两两比对,计算序列间的距离,获得一个距离矩阵.>>>邻接法(NJ)构建一个树(引导树)>>>根据引导树,渐进比对多个序列. Clustal的应用 反应序列之间两两关系;然后根据

不仅解决了大规模、多组同源序列的比对工作,同时也大大降低了运行时间,获得了较好的并行加速比(speedup),适合海量数据的分析工作. ParaAT可在不同操作系统下运行,支持多种不同的输出格式,方便后续相关的生物信息学分析(如用于检测自然选择压力的K

在 Tools 中选择 Align Two Sequence 进行双序列的比对,结果一目了然,mismatch 都被红色标注清楚,双击想看的位置可进入到对话框中,方便找到突变的位置. 2. 对 DNA 序列进行翻译 先发个软件的链接,需要的小伙伴可
网友:孙津俟:仅仅迈了一步,便直接抵达宫殿深处的其中一小院内的弃湖君面前。
网友:马奥: 狂妄!霸道!疯狂!
网友:刘誉:“谢岳父大人了。
网友:李艴渭:又不敢大意的对着兕首穷奇的脑袋一通猛扎。
网友:王狩蒙:我就无法和勾陈帝君相比。
- 上一篇:斗鱼鱼皇 斗鱼鱼皇现实干什么的_斗鱼鱼皇和银子闹翻了
- 下一篇:返回列表



